Exemples d'analyse en composantes principales
Nuage de points dans $\mathbb{R}^2$
Voir le
Notebook IPython de cet exemple. Pour afficher les graphiques avec python, exécutez la commande pl.show() après chaque suite de commande pl. Comme exercice, générez une matrice X de votre choix de taille N x 10, avec N>10. Calculer et visualisez les deux (ou trois) premières composantes principales.
NIH Blueprint Non-Human Primate (NHP) Atlas
Exemple avec données du NIH Blueprint Non-Human Primate (NHP) Atlas.
Les données originales peuvent être téléchargées à partir d'ici (cliquez sur download this data en bas de la page).
Les fichiers dont vous aurez besoin pour l'exemple
- Fichier Matlab (.mat) avec les données formattées. Le fichier contient une matrice de d'expression de gènes, une description des gènes et une description des conditions d'observation. (Fichier volumineux)
- Script Matlab pour l'analyse en composantes principales.
Pour lancer Matlab depuis les terminaux, il faut d'abord créer un alias. D'abord il faut trouver le chemin de l'application Matlab, par exemple /usr/local/bin/matlab ou /usr/local/bin/matlab2012a. Une fois localisé, éditer (ou créer s'il n'existe pas) le fichier ~/.profile
et ajouter les lignes suivantes# Set PATH variable export PATH=/usr/local/bin/matlab:$PATH
(Remplacer /usr/local/bin/matlab par votre chemin de Matlab.) Il faudra redémarrer le terminal de commande pour que le chemin soit effectif.
Chargement des données. La variable X contient les données d'expression de gène pour 2000 gènes (un gène par colonne) et 1878 conditions (une condition par ligne). La variable Probes contient les informations sur chaque gène et la variable col est une matrice de couleurs RGB correspondant au conditions (ici la structure du cerveau où le l'expression des gènes a été mesurée).
>> %% import data
>> clear; % clear all workspace variables
>>
>> % Load the data
>> load expressiondata;
>>
>> % list of variables in the workspace
>> whos
Name Size Bytes Class Attributes
Probes 2000x15 2368794 dataset
X 1878x2000 30048000 double
col 1878x3 45072 double
>>
>> % plot the gene expression matrix as image
>> figure(1); clf;
>> imagesc(X)
>> xlabel('gene')
>> ylabel('condition')
On performe la décomposition en valeurs singulières, et on calcules les composantes principales P = X*V
>> %% Perform SVD >> [U,S,V] = svd(X); >> P = X*V; % Principal componentsOn affiche les trois premières colonnes de la matrice X.
%% Plot first three components of X
figure(2); clf;
scatter3(X(:,1),X(:,2),X(:,3),24,col,'filled')
xlabel('Probes.geneid(1)')
ylabel('Probes.geneid(2)')
zlabel('Probes.geneid(3)')
On affiche les trois première composantes principales (les trois premières colonnes de P).
>> figure(3); clf;
>> scatter3(P(:,1),P(:,2),P(:,3),24,col,'filled')
>> xlabel('premiere composante')
>> ylabel('deuxieme composante')
>> zlabel('troisieme composante')
Exemple d'approximation de matrice par SVD
Les fichiers dont vous aurez besoin
Avec python:
>>> from numpy import *
>>>
>>> im = genfromtxt('image.csv')
>>>
>>> # Decomposition en valeurs singulières
... U, s, Vs = linalg.svd(im, full_matrices=True)
>>>
>>> # Dimension des matrices
... U.shape
(115, 115)
>>> s.shape
(115,)
>>> Vs.shape
(130, 130)
>>>
>>> # s est un vecteur, on reconstruit une matrice diagonale
... S = zeros((115,130))
>>> S[:115,:115] = diag(s)
>>>
>>> r = array([1, 2, 4, 8, 16, 32, 64, 115])
>>> for k in r:
... SS = zeros((115,130))
... SS[:k,:k] = diag(s[:k])
...
... # matrice de rang r
... MM = dot(U,dot(SS,Vs))
...
... print 'r = {r}, rang de MM = {rang}'.format(r=k,rang=linalg.matrix_rank(MM))
...
... fname = 'svd_rank_{r}.csv'.format(r=k)
... savetxt(fname,MM)
...
r = 1, rang de MM = 2
r = 2, rang de MM = 3
r = 4, rang de MM = 5
r = 8, rang de MM = 9
r = 16, rang de MM = 17
r = 32, rang de MM = 33
r = 64, rang de MM = 64
r = 115, rang de MM = 115




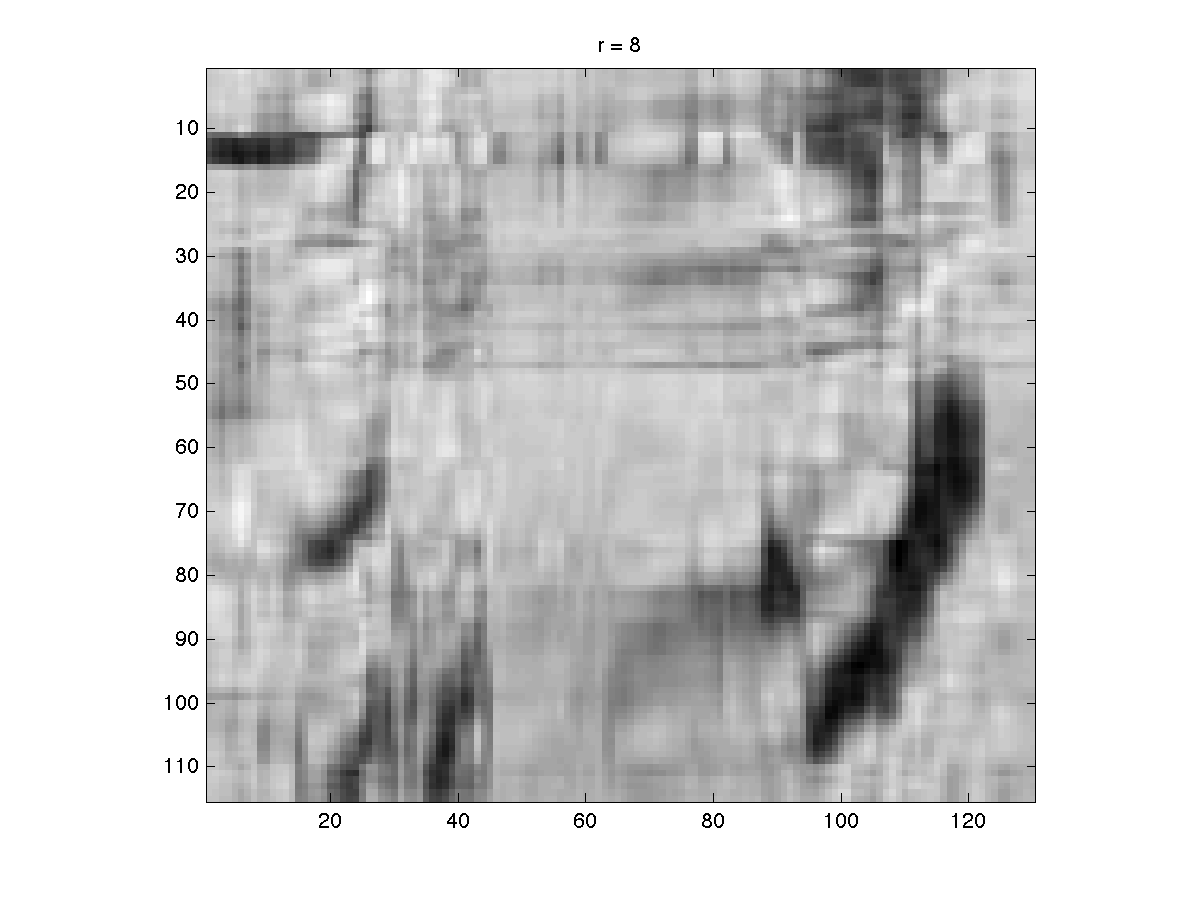




Pour visualiser par exemple la matrice de rang 32 avec gnuplot
svd_lowrank$ gnuplot gnuplot> set palette gray gnuplot> set yrange [0:114] reverse gnuplot> set xrange [0:129] gnuplot> plot "svd_rank_32.csv" matrix w image